在机器学习和数据分析中,数据的维度常常是一个让人头疼的问题。
想象一下,你面前有一张包含成千上万列特征的表格,每一列都可能是一个重要的信息源,但同时也会让计算变得异常复杂。
这时候,降维技术就派上用场了!它可以帮助我们把高维数据“瘦身”成低维数据,同时尽可能保留有用的信息。
今天,介绍几种常见的降维方法的原理和应用方式。
1. k近邻学习:寻找“邻居”的智慧
k近邻学习(k-Nearest Neighbors,简称k-NN)是一种简单而强大的算法,虽然它本身主要用于分类和回归,但它的思想也可以启发我们进行降维。
k近邻的核心思想是:如果一个数据点周围的k个邻居都属于某个类别,那么这个数据点很可能也属于这个类别。
在降维的语境中,我们可以利用这种“邻居关系”来简化数据。
假设我们有一个高维数据集,每个数据点都有很多特征,k近邻学习会计算每个点与其他点之间的距离,找到距离最近的k个点(邻居)。
通过这种方式,我们可以构建一个基于“邻居关系”的低维表示。
例如,如果两个点在高维空间中有很多邻居是相同的,那么它们在低维空间中也应该靠近。
虽然k近邻本身不是一种降维算法,但我们可以用它来构建一个简单的降维示例。
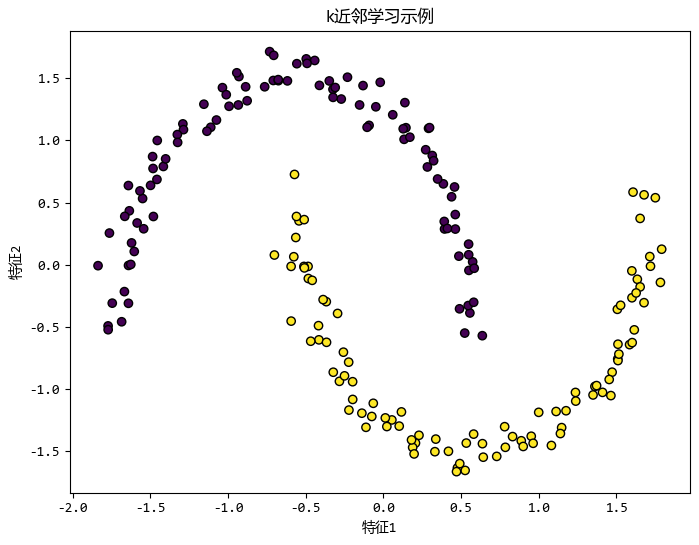
这里我们用scikit-learn的KNeighborsClassifier来演示如何通过邻居关系来简化数据。
import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
from sklearn.datasets import make_moons
from sklearn.preprocessing import StandardScaler
# 创建一个简单的二维数据集
X, y = make_moons(n_samples=200, noise=0.05, random_state=42)
scaler = StandardScaler()
X = scaler.fit_transform(X)
# 使用k近邻分类器
knn = KNeighborsClassifier(n_neighbors=5)
knn.fit(X, y)
# 绘制数据和邻居关系
plt.figure(figsize=(8, 6))
plt.scatter(X[:, 0], X[:, 1], c=y, cmap="viridis", edgecolor="k")
plt.title("k近邻学习示例")
plt.xlabel("特征1")
plt.ylabel("特征2")
plt.show()
2. 低维嵌入:把高维数据“塞”进低维空间
低维嵌入是一种将高维数据映射到低维空间的技术,同时尽量保留数据的结构和关系。
它的目标是让相似的数据点在低维空间中依然保持相似,而不同的数据点则尽可能分开。
低维嵌入的核心是通过某种映射函数,将高维数据点转换为低维数据点。
这个映射函数通常是通过优化一个目标函数来学习的,比如最小化数据点之间的距离差异。
常见的低维嵌入方法包括t-SNE和UMAP。
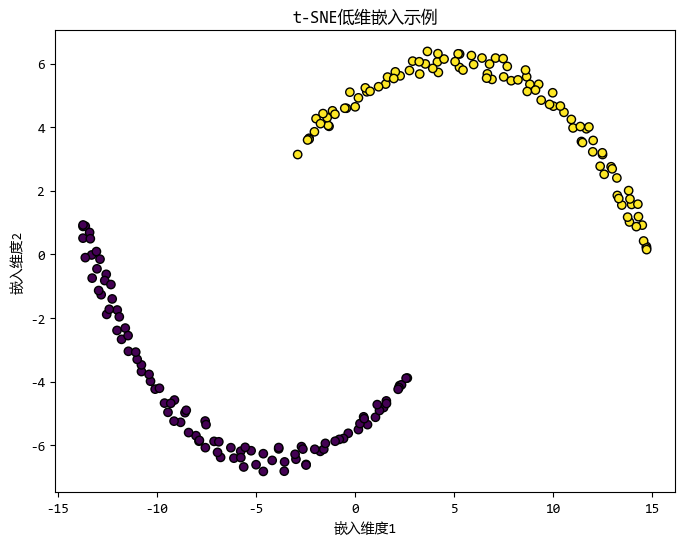
下面我们用scikit-learn的t-SNE来演示低维嵌入的效果。
from sklearn.manifold import TSNE
# 创建一个高维数据集
X, y = make_moons(n_samples=200, noise=0.05, random_state=42)
X = scaler.fit_transform(X)
# 使用t-SNE进行低维嵌入
tsne = TSNE(n_components=2, random_state=42)
X_embedded = tsne.fit_transform(X)
# 绘制嵌入后的数据
plt.figure(figsize=(8, 6))
plt.scatter(X_embedded[:, 0], X_embedded[:, 1], c=y, cmap="viridis", edgecolor="k")
plt.title("t-SNE低维嵌入示例")
plt.xlabel("嵌入维度1")
plt.ylabel("嵌入维度2")
plt.show()
3. 主成分分析(PCA):找到最重要的“方向”
主成分分析(PCA)是一种经典的降维方法,它的目标是找到数据中最重要的“方向”,并沿着这些方向对数据进行投影,从而降低维度。
PCA通过计算数据的协方差矩阵,找到数据的主成分(即方差最大的方向),这些主成分是数据中最“重要”的特征方向。
通过保留前几个主成分,我们可以将高维数据投影到低维空间,同时尽可能保留原始数据的方差。
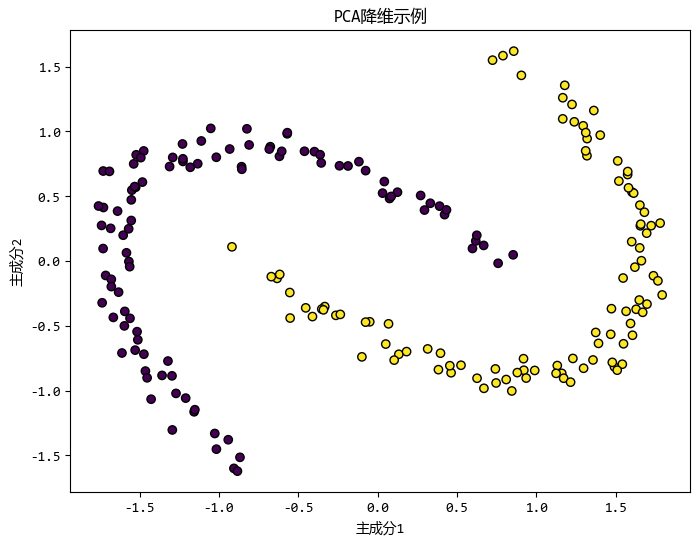
示例代码如下:
from sklearn.decomposition import PCA
# 创建一个高维数据集
X, y = make_moons(n_samples=200, noise=0.05, random_state=42)
X = scaler.fit_transform(X)
# 使用PCA进行降维
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X)
# 绘制降维后的数据
plt.figure(figsize=(8, 6))
plt.scatter(X_pca[:, 0], X_pca[:, 1], c=y, cmap='viridis', edgecolor='k')
plt.title("PCA降维示例")
plt.xlabel("主成分1")
plt.ylabel("主成分2")
plt.show()
4. 核化线性降维:给线性降维加上“魔法”
核化线性降维(Kernel PCA)是PCA的一种扩展,它通过引入核函数,将数据映射到一个高维的特征空间,在这个空间中进行线性降维。
核化线性降维的核心是核函数。核函数可以将数据映射到一个高维空间,使得原本线性不可分的数据在这个空间中变得线性可分。
然后,我们在高维空间中应用PCA,最后将结果映射回低维空间。
示例代码如下:
from sklearn.decomposition import KernelPCA
# 创建一个高维数据集
X, y = make_moons(n_samples=200, noise=0.05, random_state=42)
X = scaler.fit_transform(X)
# 使用核化PCA进行降维
kpca = KernelPCA(n_components=2, kernel='rbf', gamma=15)
X_kpca = kpca.fit_transform(X)
# 绘制降维后的数据
plt.figure(figsize=(8, 6))
plt.scatter(X_kpca[:, 0], X_kpca[:, 1], c=y, cmap='viridis', edgecolor='k')
plt.title("核化PCA降维示例")
plt.xlabel("核化主成分1")
plt.ylabel("核化主成分2")
plt.show()
5. 流形学习:沿着数据的“形状”降维
流形学习是一种基于流形假设的降维方法,它认为高维数据实际上是一个低维流形的嵌入。
流形学习的目标是恢复这个低维流形的结构。
流形学习的核心是假设数据在高维空间中是沿着某个低维流形分布的。
通过学习这个流形的结构,我们可以将数据映射到低维空间,同时保留数据的局部结构。
下面用scikit-learn的Isomap来演示流形学习的效果:
from sklearn.manifold import Isomap
# 创建一个高维数据集
X, y = make_moons(n_samples=200, noise=0.05, random_state=42)
X = scaler.fit_transform(X)
# 使用Isomap进行流形学习
isomap = Isomap(n_components=2)
X_isomap = isomap.fit_transform(X)
# 绘制降维后的数据
plt.figure(figsize=(8, 6))
plt.scatter(X_isomap[:, 0], X_isomap[:, 1], c=y, cmap='viridis', edgecolor='k')
plt.title("Isomap流形学习示例")
plt.xlabel("流形维度1")
plt.ylabel("流形维度2")
plt.show()
6. 总结
降维技术在数据分析和机器学习中扮演着重要的角色。
实际应用时,建议:
- 线性数据优先尝试PCA
- 可视化需求首选t-SNE
- 特征工程推荐Kernel PCA
- 地理类数据适合MDS
- 复杂流形使用Isomap/LLE
通过灵活组合这些方法,可以有效提升模型性能,发现数据背后的本质结构。
