深度学习课题总结
课题目标:
基于 3D 重建的早期结肠肿瘤分割。
背景:
结肠癌是全球 第三大常见致死癌症,每年新增患者 100-200 万,死亡约 70 万。
结肠镜是结肠癌筛查的 金标准,目标是检测潜在癌变的息肉:
- 高危息肉: 大小超过 1 cm,被 AGA 协会视为高风险肿瘤
-
恶变概率高: 大息肉最终发展为肿瘤的比例显著
然而,当前诊断依赖于医生经验,存在较高误诊率。一项研究显示,医生对息肉大小估计错误的概率高达 50%,影响风险评估和治疗方案。
动机:
- 潜在威胁: 早期结肠癌源于粘膜病变(如腺瘤性息肉),由于缺乏早期诊断,许多患者发现时已处于晚期(Stage IV),治疗效果有限。
- 可行预防: 提高息肉检测率可显著降低癌症发生率,每 增加 1% 的检测率,可减少 3% 的结肠癌发病率。
目标:
开发基于 深度学习的 3D 重建框架,准确分割结肠镜图像中的可疑肿瘤,实现:
- 对息肉 不规则形状与模糊边界的风险量化
- 为息肉活检决策提供 客观支持,减少医生主观判断偏差
- 对高风险病变区域 最大程度降低漏诊率
挑战:
- 息肉形状高度不规则
- 息肉边界模糊,分割困难
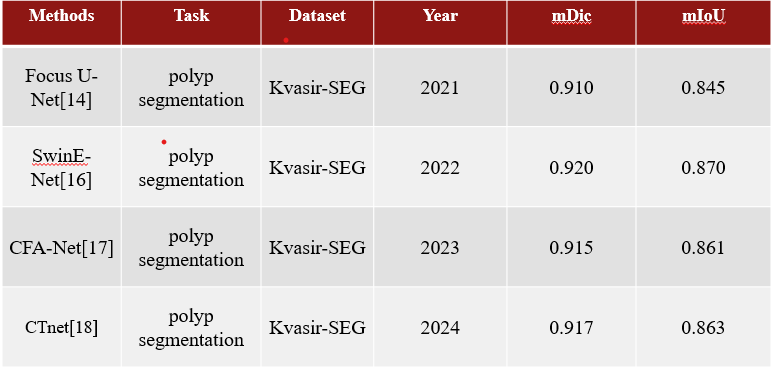
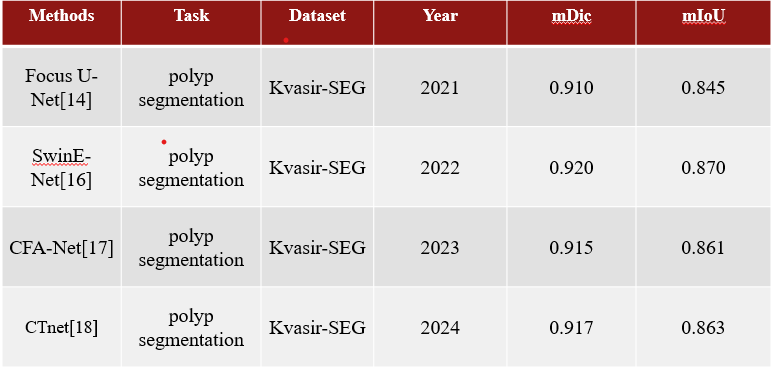
相关工作:
实验:
-
实验流程设计:
使用 U-Net 对图像进行 2D segmentation
使用 NeRF 进行 3D reconstruction
将重建结果投影回 2D,与 2D segmentation 结果比较,突出 3D reconstruction 的必要性 -
数据集:
Kvasir-SEG: 1000 张息肉图片,提供像素级 segmentation masks
LDPolypVideo:160 个结肠镜视频,40266 帧(560×480)带息肉标注的图片,提供原始视频文件。 -
实验过程:
3.1 Kvasir-SEG 2D 分割:
模型:U-Net
Dice = 0.9082,IoU = 0.8684
3.2 Kvasir-SEG 3D 重建:
结果不理想,原因:Kvasir-SEG 图片非连续帧,缺少相机位姿信息,导致 3D reconstruction 失败。
3.3 LDPolypVideo 数据集:
使用 26000+ 张图片训练 U-Net
训练集结果:Dice = 0.9429,IoU = 0.90402
3.4 COLMAP 3D 重建:
对单个视频进行 feature extraction、matching 和 reconstruction
成功生成 3D 点云
课题结果:
由于曾经在期末答辩中,息肉检测与分割课题被评审认为研究已趋成熟,教授决定调整你的研究方向,指派你参与 MIDOG 2025 竞赛,作为下一阶段主要课题。
